
甘蔗(Saccharum spp.)是一种古老的作物,据信约在1万年前在新几内卜亚被驯化,在世界近代史上,甘蔗的种植极大地促进了世界范围内人口的迁徙,包括奴隶贸易和劳工,对人类历史产生深远的影响。甘蔗也是世界上最重要的糖料作物和能源作物,贡献了全球80%的糖和40%的乙醇,年产值超过900亿美元。全球目前有超过100个国家种植甘蔗。一般而言,甘蔗属包括高贵种(Saccharum officinarum)、中国种(Saccharum sinense)、印度种(Saccharum barberi)、食穗种(Saccharum edule)、大茎野生种(Saccharum robustum)和割手密(Saccharum spontaneum)(Mirajkar等,2019)。其中,割手密种和大茎野生种被认为是野生种,而高贵种作为一种高糖含量的栽培种,推测是由新几内亚的大茎野生种经过人类选择而产生的(Pompidor等,2021)。它们大多数被认为是同源多倍体,基因组复杂且庞大。现代甘蔗品种源于高贵种与割手密之间的选择性种间杂交,并进行了多次回交(Lu等,1994)。现代甘蔗品种中约70-80%的染色体来自高贵种,10-23%来自割手密,5-17%则是这两种之间重组的结果(Aitken等,2014;D'Hont等,1996;Tu等,2009),使现代栽培甘蔗的基因组成为栽培作物中最复杂的之一。此外,甘蔗与河八王、蔗茅、斑茅和芒属共同构成了“甘蔗复合体”, 这些近缘属野生种是拓宽甘蔗遗传基础的重要种质资源。这一密切相关的杂交群体被认为与甘蔗的起源有关,突显了其对遗传研究和育种计划的重要性(D'Hont等,1996)。
由于甘蔗极高程度的多倍体特性,其具有所有大宗作物中最为复杂的基因组。在过去的20多年里,巴西、法国、中国、澳大利亚、美国、南非、印度、泰国和哥伦比亚等,相继投入力量开展了甘蔗基因组领域的攻关,并取得了一定的成绩。近年来,随着基因测序和基因组组装技术的快速发展,甘蔗基因组的解析工作已经开始。张积森教授团队于2018年发布割手密AP85-441的基因组(Zhang等,2018),2022年发布了割手密Np-X基因组(Zhang等,2022),2024年发布了现代栽培种XTT22的基因组(Zhang等,2024),以及其他团队发布的现代栽培种R570基因组(Healey等,2024)和现代栽培种ZZ1基因组(Bao等,2024)。与甘蔗研究相关的转录组数据也丰富,涵盖了各种植物组织、发育阶段、昼夜节律等(Ma等,2020;Zhang等,2021)。为了更好地利用近年来生成的大量甘蔗基因组、转录组及其他数据,需要一个平台来组织和共享这些数据,在前人的研究中开发了多个甘蔗基因组数据库,如SymGRASS(http://symgrass.dyndns.org)(Belarmino等,2013),该数据库编目了甘蔗的同源基因,以及甘蔗miRNAs数据库(https://sysbiol.ib.unicamp.br/SCmiRNA/)(Zanca等,2010),整合了甘蔗miRNA初级前体和靶标的信息。尽管这些数据库非常有价值,但通常设计为要么收集全面的甘蔗基因组数据,要么集中于特定的组学领域。
为了使用户更好地利用这些测序数据,广西大学张积森教授团队开发了(ScDB,https://sugarcane.gxu.edu.cn/scdb/),于2024年8月30日,在Plant Biotechnology Journal发表了题为“ScDB: A comprehensive database dedicated to Saccharum, facilitating functional genomics and molecular biology studies in sugarcane”的简短通讯,这是一个综合性和多功能平台,提供多种实用的分析工具,用户能够轻松查找感兴趣的基因,获取功能注释和序列等信息,分析和可视化基因表达模式,通过共表达网络和蛋白质相互作用预测搜索相关基因,并进行其他分析,如引物设计和同源性块识别。

以下为数据库的用户手册:
ScDB概述结构
ScDB目前收录6种甘蔗及其近缘物种的基因组资源和组学数据,包括两个割手密种(AP85-441和Np-X)、一个高贵种(LA-Purple)、三个现代栽培种(XTT22,R570,ZZ1)和一个完整无缝的蔗茅基因组(YN2009-3)。ScDB由前端网页界面、后端应用服务器、数据库和开源分析和可视化工具组成。数据库主要功能模块有:“主页”、“基因组学”、“转录组学”、“工具”和“下载” (图1)。主页展示了ScDB的介绍、高级搜索、甘蔗属及其近缘物种的描述,以及各种工具的链接。基因组学模块包括基因搜索、共线性搜索和基因组浏览的功能。转录组学模块提供基因表达模式和共表达基因网络的搜索和可视化功能。工具模块提供了一系列数据分析和可视化的工具。用户可以从数据库的下载模块访问相关的基因组数据和注释。
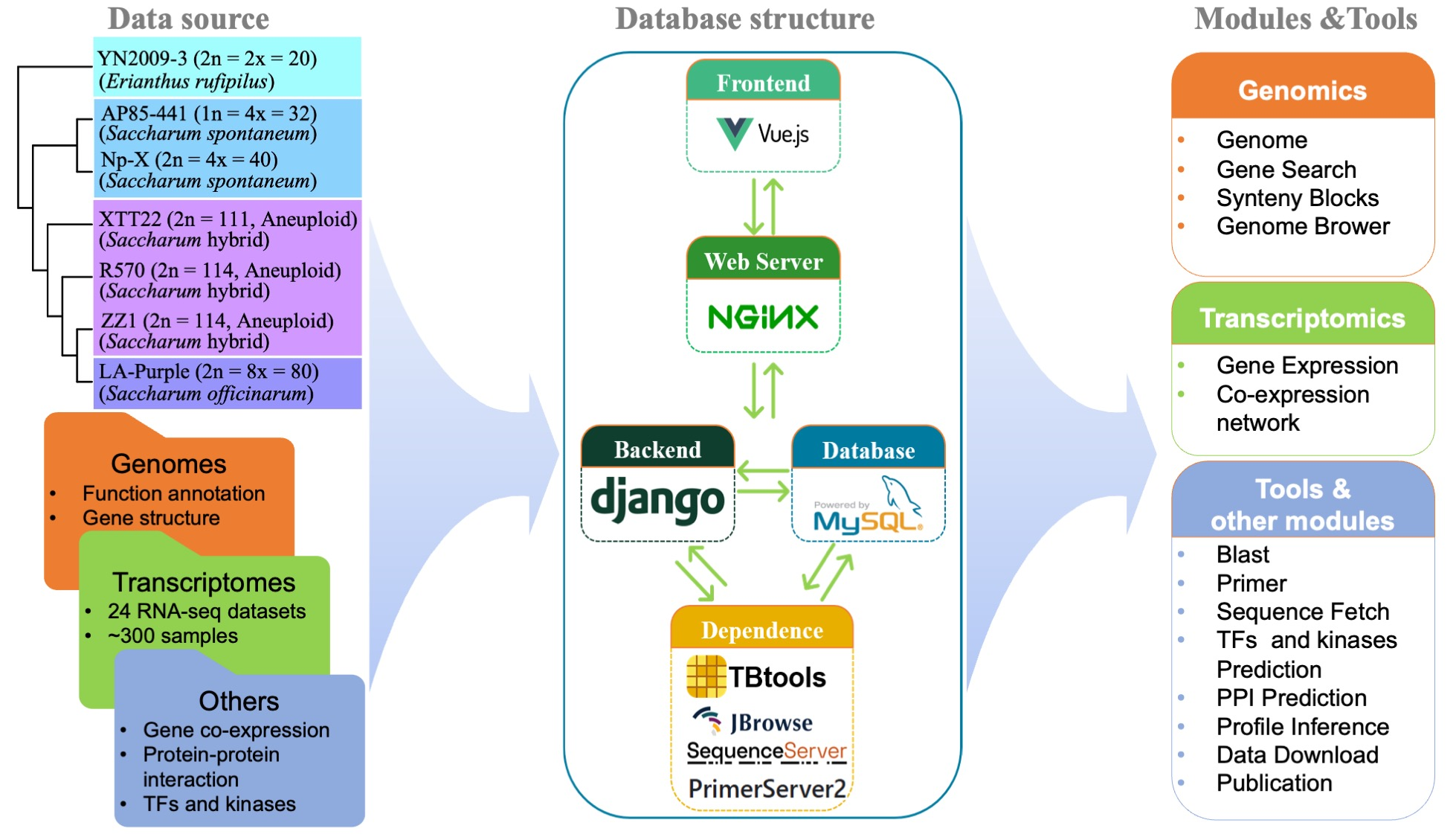
图1 ScDB技术框架
基因组学模块
基因组页面(Genome ,https://sugarcane.gxu.edu.cn/scdb/genomics/genome)展示数据库收录物种的基因组基本信息,点击基因组名称会链接到一个单独的页面,显示详细的基因组信息、相关论文和染色体水平的结构注释细节,包括miRNA、tRNA等。基因搜索页面(Gene Search ,https://sugarcane.gxu.edu.cn/scdb/genomics/gene_search)用户可以通过基因ID或染色体位置使用基因搜索功能搜索基因。基因搜索结果页面包含基本基因信息、注释结果(包括KEGG通路和GO功能注释)。在首页的高级搜索和基因搜索页面搜索到的基因点击链接即可进入基因详情页面,包含该基因的染色体位置信息、功能注释、表达量柱形图、同源基因以及CDS、蛋白质和上下游序列(图2A)。在同源基因搜索页面(https://sugarcane.gxu.edu.cn/scdb/genomics/orthologous)用户可以通过基因ID搜索以获取其他甘蔗基因组和拟南芥和水稻中同源基因的信息。支持的物种及基因ID如页面案例所示。共线性页面(https://sugarcane.gxu.edu.cn/scdb/genomics/synteny_blocks)用于显示甘蔗基因组之间的共线性(图2B)。包含两个工具,在基因共线性中,通过基因ID检索其在其他物种中的共线性基因。在染色体共线性中,可以指定任意两个基因组的任意染色体的共线性,并且可以随意交互放大以及转向染色体。基因组浏览器(Genome Browser)页面提供访问JBrowse2(Diesh等,2023)的功能,该工具用于显示可用参考基因组组装的基因组序列、注释的基因组特征和其他基因组信息。JBrowse2允许用户添加轨道、搜索特定区域,并导出数据和图形,便于详细的基因组分析和可视化。
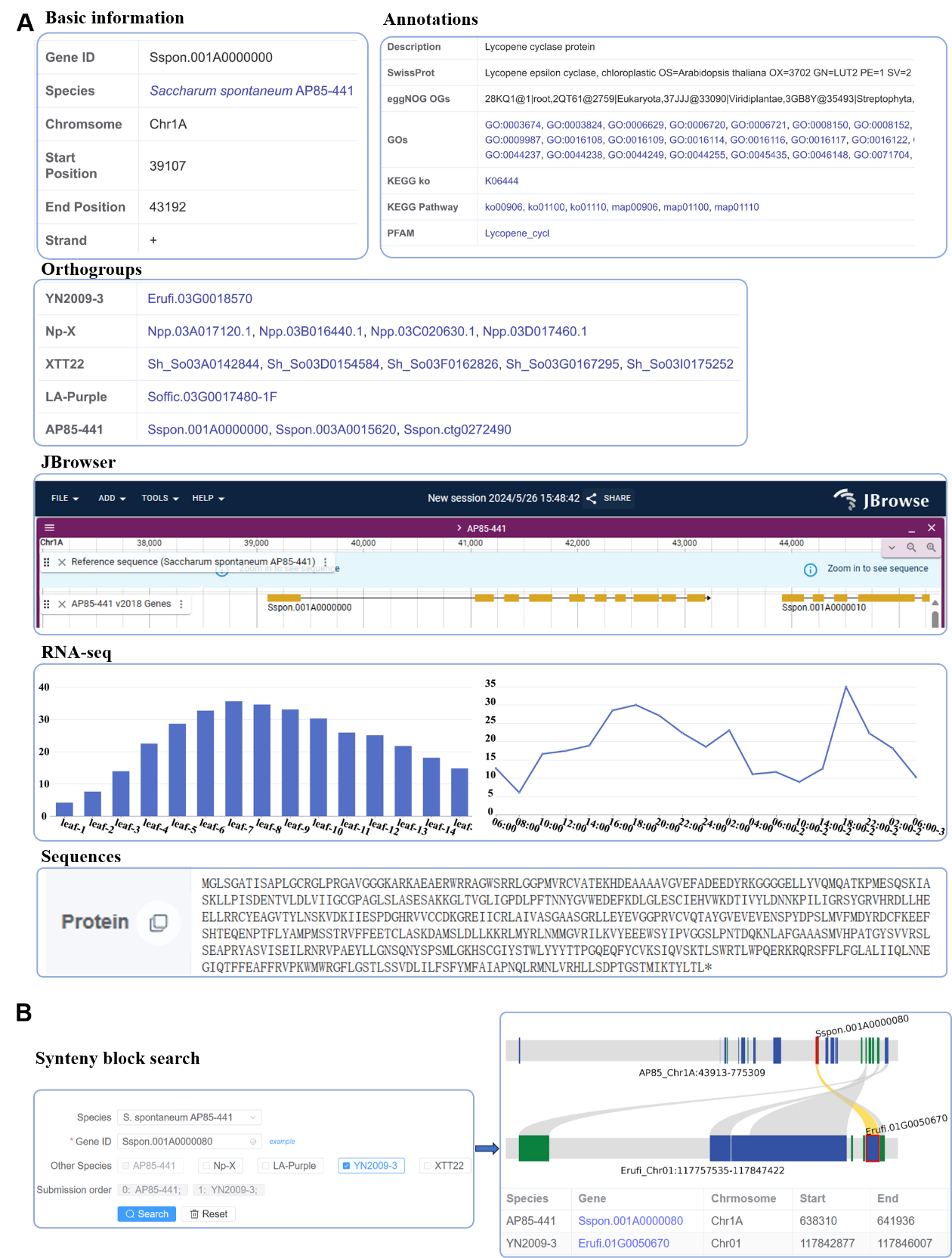
图2 基因组学模块功能
转录组学模块
该模块包括24个转录组项目,涵盖300多个甘蔗样本,约2.5 TB的数据。这些样本来自于不同的组织、发育阶段、昼夜节律或不同植物激素处理条件,详细描述可以在研究详情页面(https://sugarcane.gxu.edu.cn/scdb/transcriptomics/studies_detail)查看。
在此模块中,我们开发了基因表达热图的可视化界面(https://sugarcane.gxu.edu.cn/scdb/transcriptomics/gene_expression) (图3A)。用户可以与基因搜索相同的逻辑,通过基因ID或染色体位置搜索特定基因并分析其表达模式,从选择框中选择该物种所有的研究,任意指定热图使用TPM与FPKM作为表达量单位,并且可以自定义热图颜色方案,并导出SVG矢量图。使用WGCNA对这些转录组数据进行共表达分析,在共表达网络(Co-expression Network,https://sugarcane.gxu.edu.cn/scdb/transcriptomics/co_expression)页面中,用户可以通过基因ID检索其共表达网络的数据并可视化(图3B),并导出SVG矢量图。
使用WGCNA对这些转录组数据进行共表达分析,在共表达网络(Co-expression Network,https://sugarcane.gxu.edu.cn/scdb/transcriptomics/co_expression)页面中,用户可以通过基因ID检索其共表达网络的数据并可视化(图3B),并导出SVG矢量图。
 图3 转录组学模块功能
图3 转录组学模块功能
工具模块
目前,ScDB的工具模块包括六个分析处理工具:Blast(https://sugarcane.gxu.edu.cn/scdb/tools/blast)、引物(Primer ,https://sugarcane.gxu.edu.cn/scdb/tools/primer)、序列获取(Sequence Fetch,https://sugarcane.gxu.edu.cn/scdb/tools/seq_fetch)、转录因子查询(Transcription Factors,https://sugarcane.gxu.edu.cn/scdb/tools/tf)、蛋白质相互作用预测(Protein Interaction Network,https://sugarcane.gxu.edu.cn/scdb/tools/ppi)和motif预测(Profile Inference,https://sugarcane.gxu.edu.cn/scdb/tools/profile_inference)。这些工具对研究人员非常有价值,允许他们将自己的研究与ScDB整合以获得所需结果。
Blast工具是使用SequenceServer3(Priyam等,2019)软件构建的,用户可以粘贴查询序列(DNA或蛋白质)或上传包含查询序列的FASTA格式文件,然后选择Blast参数,包括最大比对数和E-value,从而在甘蔗基因组中查找相似基因。
引物工具基于PrimerServer2(https://github.com/billzt/PrimerServer2)(Zhu等,2017)提供针对甘蔗基因组中特定染色体位置和靶序列的高通量引物设计。输入格式为染色体名,开始位置,以及区间大小,并以空格分隔,程序就可以在指定区间内按要求设计引物。还可以进行引物特异性检查,搜索可能的扩增子并过滤出特定引物,确保引物唯一结合于靶序列。
在蛋白质-蛋白质相互作用(PPI)工具中,用户可以通过基因ID搜索特定基因的蛋白质相互作用网络。结果以表格形式呈现,可以保存为CSV文件,也可以可视化为交互式网络图,后者也可以保存为SVG图像(图4A)。
在转录因子工具页面中,我们使用iTAK软件(Zheng等,2016)预测了数据库收录基因组的转录因子家族和激酶家族。我用户在搜索框输入基因组检索其所在家族,点击任何转录因子家族或激酶家族的名称,以查看该家族中包含的所有基因列表(图4B)。
在motif预测工具中,支持用户输入基因ID,基因名和蛋白质序列,预测它们可能结合的启动子基序(图4C),提供motif图片及meme文件下载,从而用于预测它们可能调控的基因。
下载模块和出版模块
下载页面提供与甘蔗属基因组相关的各种数据集的访问。用户可以下载基因组组装(FASTA)、基因模型序列(FASTA)、基因编码序列(FASTA)、蛋白质序列(FASTA)、基因结构注释(GFF3)和其他相关资源,以支持甘蔗的全面基因组研究和分析。出版页面列出了与甘蔗相关的各种研究论文和文章。这些出版物涵盖了基因组测序、功能基因组学和分子生物学等多个主题,为甘蔗研究提供了宝贵的见解和进展。
案例研究:生长调节因子的识别与特征分析
为了展示ScDB的详细功能,我们进行了一项案例研究,模拟实际研究场景。我们在AP85-441基因组中识别了一个GRF基因,并进行了相关的生物信息学分析。
生长调节因子(GRFs)是植物特有的转录因子,在调控植物生长和发育中发挥着重要作用(Kuijt等,2014;Vercruyssen等,2015)。在我们之前对割手密种基因组的研究中,我们识别了一个GRF基因GRF7,促进甘蔗的叶片生长和扩展(Wang等,2024)。因此,我们选择GRF7作为示例来测试ScDB的各种功能。
首先,我们使用水稻GRF蛋白质序列作为查询序列,通过ScDB工具模块中的Blast功能在AP85-441基因组中执行同源基因搜索(图5A)。接下来,为了获得最佳匹配基因(总分最高)的详细信息,我们在基因组模块中使用基因搜索功能搜索基因ID “Sspon.005B0003552”。在结果页面(图5B),用户可以获得该基因的详细信息。该基因位于5B染色体上,包含两个保守域QLQ和WRC,这些域对GRF基因至关重要(Kim和Kende,2004)。该基因的表达模式也在结果页面上显示。它在成熟茎和叶基部的表达水平最高。此外,该基因似乎对昼夜转变有反应,表达水平在上午10:00后上升,晚上10:00后下降(图5C)。接下来,我们使用共表达网络功能识别了与其共表达的262个基因(图5D)。此外,利用工具模块中的转录因子工具,我们识别了AP85-441基因组中的所有GRF家族基因。然后,我们为这些基因生成了基因表达热图,显示所有GRF基因表现出相似的表达模式(图5E)。由于GRF基因是转录因子,用户可以利用工具模块中的谱系推断工具预测其可能结合的启动子区域的基序(图5F)。
讨论
虽然 ScDB 提供了一个可用的平台,但未来仍有改进的空间。多倍体基因组存在多条同源染色体和单倍型,这给数据库准确呈现数据带来了挑战。由于存在多个等位基因,ScDB 目前的同源可视化模块生成的图像过于拥挤,难以直观地获取信息。此外,转录组学模块生成的热图不能有效显示相关等位基因的表达水平。我们正在积极寻求解决这些问题的方法,预计很快就能解决。随着技术的进步,与甘蔗研究相关的数据集将越来越丰富。我们的数据库将不断更新,为甘蔗数据采集和分析提供一个更强大、更可持续的平台。
该论文由广西大学生命科学与技术学院博士生陈思远和冯晓溪为论文共同第一作者,广西大学亚热带农业生物资源保护与利用国家重点实验室张积森教授为论文的通讯作者。福建农林大学海峡联合研究院基因组中心唐海宝教授、明瑞光教授和广西大学亚热带农业生物资源保护与利用国家重点实验室张木清教授、陈宝善教授和姚伟教授参与了本研究。同时,本研究还得到了华南农业大学的陈程杰副研究员和暨南大学李嘉威博士后在数据分析等方面的重要的指导和帮助。该项目得到了国家自然科学基金、国家重点研发计划的支持。张积森团队长期致力于甘蔗基因组与分子育种研究,近年来在Nature Genetics(Zhang等,2018;Zhang等,2022;Zhang等,2024)、Nature Plant(Wang等,2023)、The Plant Journal(Zhang等,2020;Jiang等,2023)、Plant Biotechnology Journal(Zhang等,2019;Yang等,2019)等权威期刊发表相关论文,在甘蔗基因组与分子育种研究中取得许多重要进展。
文章引用
Chen, S., Feng, X., Zhang, Z., Hua, X., Zhang, Q., Chen, C., et al. (2024) ScDB: A comprehensive database dedicated to Saccharum, facilitating functional genomics and molecular biology studies in sugarcane. Plant Biotechnology Journal, pbi.14457. https://doi.org/10.1111/pbi.14457
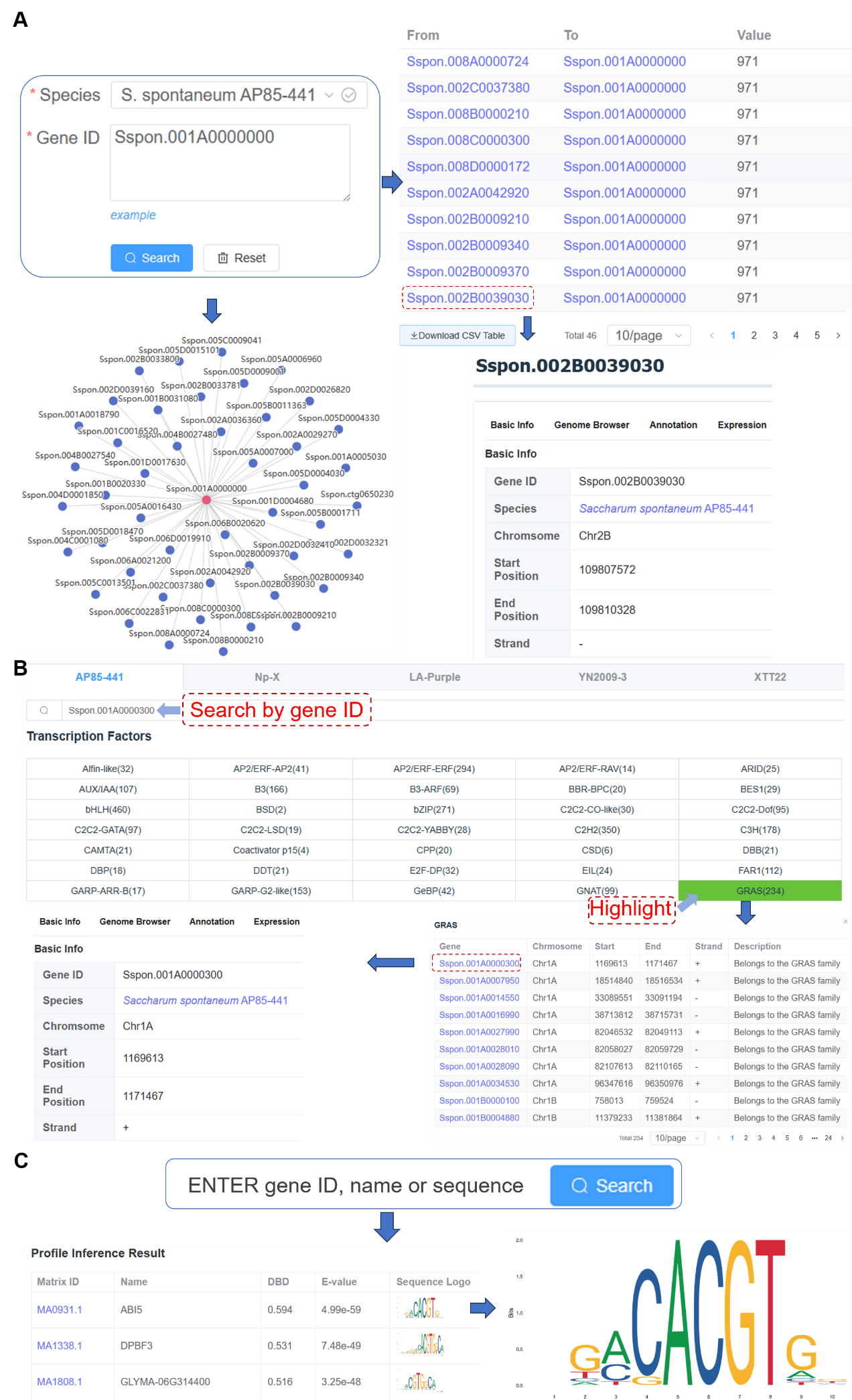
图4 工具模块功能
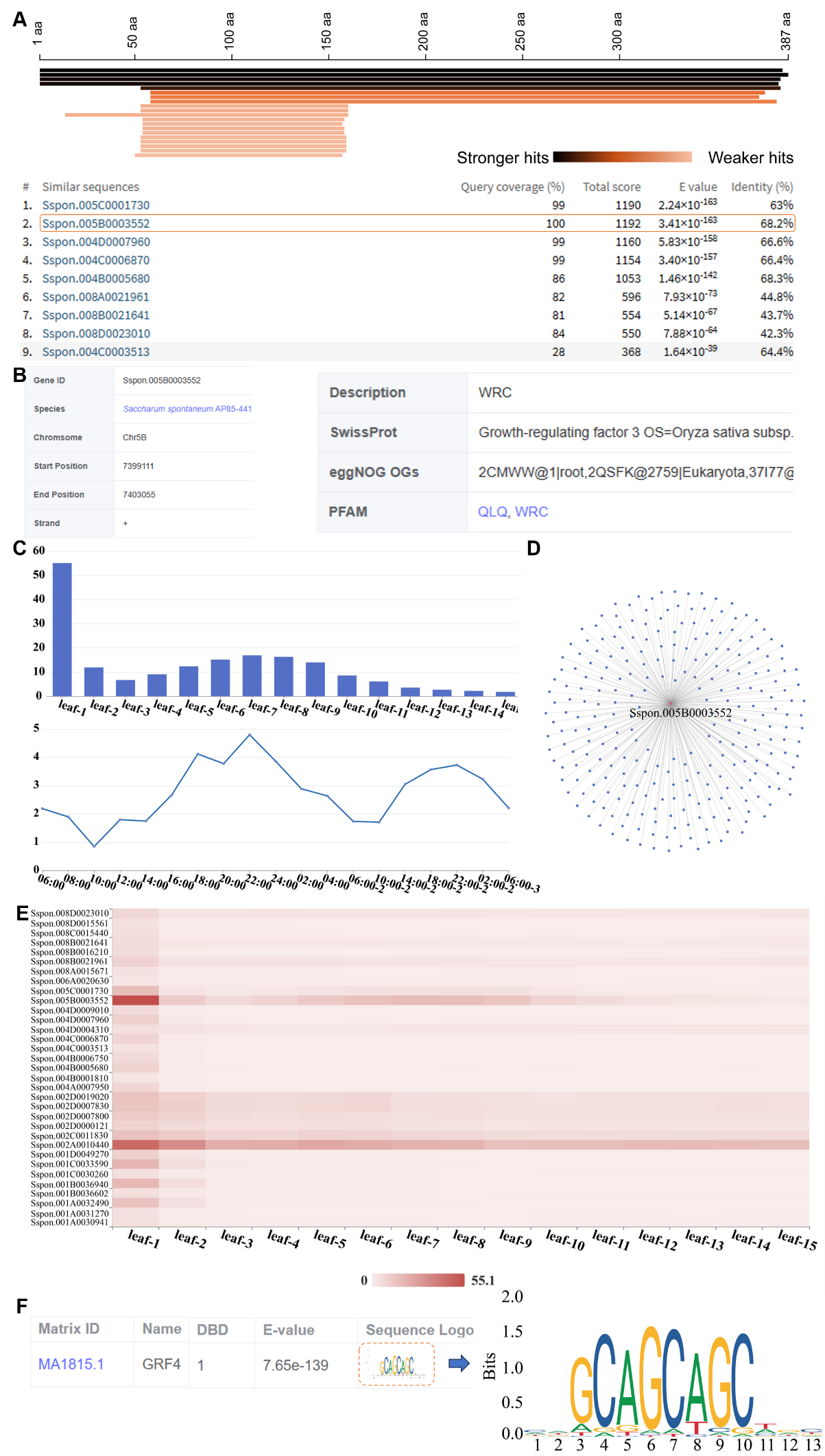
图5 ScDB 中 GRF 基因的分析结果说明。
材料和方法
数据库实现
ScDB 数据库部署在一个基于CentOS的Linux服务器上,利用Nginx 网络服务器 (https://nginx.org/)获得强大而高效的性能。数据存储和管理由 MySQL 数据库管理系统 (https://www.mysql.com/)处理,以确保可靠和可扩展的数据处理能力。后台功能由 Django (https://www.djangoproject.com/)支持,为服务器端操作提供了坚实的基础,而前端则使用 Vue.js (https://vuejs.org/)开发,结合Element Plus(https://cn.element-plus.org/)和Apache ECharts组件(https://echarts.apache.org/zh/index.html)组件库为用户提供了直观、友好的界面。该网站在 Firefox、Google Chrome 和 Safari 等主要网络浏览器上进行了广泛的测试。这些测试显示了网站的稳定性能,并确保了在各种平台上的兼容性。
基因组数据和分析
ScDB 整合了甘蔗基因组数据,包括基因组、基因序列和结构注释。R570与ZZ1来源于文章公开数据,其他物种数据均来自张积森团队。我们使用 EggNOG-mapper (http://eggnog-mapper.embl.de/)和 KOBAS 3.0(http://kobas.cbi.pku.edu.cn/)在全基因组水平分别对照 EggNOG 5(http://eggnog5.embl.de/)和 KEGG 数据库对每个物种的预测基因进行了注释,以检索 GO 术语和 KEGG(Bu 等,2021 年;Huerta-Cepas 等,2019 年)。
共表达网络构建
为了确定五种蔗糖树及其近缘种的共表达基因,我们构建了共表达网络。我们使用 R 软件包 WGCNA(版本 1.72-1)(Langfelder 和 Horvath,2008 年)构建了共表达网络。这种方法利用加权相关矩阵来评估不同样本中基因表达模式之间的关系。
基因组共线性与同源基因分析
基因组共线性和同源基因为了解物种的进化信息和功能保护提供了宝贵的信息。同源基因使用 OrthoFinder(2.5.5 版)(Emms 和 Kelly,2019 年)进行鉴定。使用 MCScan(Tang 等人,2008 年)以默认参数进行分析。可视化使用 TBtools(Chen 等,2023 年)实现。
转录因子和激酶分析
转录因子和激酶对调控各种生物过程(包括基因表达和信号转导)至关重要。我们使用 iTAK(2.0.2 版)(Zheng 等人,2016 年)鉴定了数据库包含物种的转录因子家族和激酶家族,该软件是鉴定和分类植物转录因子和蛋白激酶的综合工具。
蛋白质互作分析
蛋白质互作(PPI)分析是了解蛋白质之间功能关联的重要方法。我们使用 STRING (https://string-db.org/)(Szklarczyk et al., 2022)确定了 PPI 网络。