2024年8月18日,广西大学陈玲玲教授团队在Plant Communications发表了题为“Direct RNA sequencing in plants: practical applications and future perspectives”的综述文章。该综述总结了近年来以RNA直接测序(Directed RNA sequencing, DRS)为主要技术手段剖析植物转录组复杂性和多样性的研究进展,系统阐述了DRS相较于传统RNA-seq的优势,针对植物DRS数据分析提出了详细的流程和工具。此外,文章还深入探讨了DRS技术在植物研究中面临的挑战,展望了其在未来植物生物学研究中的广阔前景。

相较于传统的RNA-seq需要经过cDNA合成和PCR扩增等步骤,Oxford Nanopore Technologies (ONT) 的RNA直接测序技术(DRS)能够直接对原始RNA分子进行全长测序,避免引入测序偏差并保留RNA的完整性和修饰信息(图1)。这使得DRS在发现新基因、可变剪接事件、非编码RNA以及RNA修饰方面具有显著优势。

图1. 直接RNA测序(DRS)与全转录组测序文库构建流程的比较(A),以及DRS在植物研究中的主要应用(B)
本综述涵盖了DRS技术在植物转录组研究中的主要进展,重点探讨了转录组复杂性、新基因和可变剪接体的发现、非编码RNA(lncRNAs)和环状RNA(circRNAs)的鉴定、植物中融合转录本的研究现状、poly(A)尾的功能及RNA碱基修饰与其他RNA特征之间的关系,特别强调了该技术在揭示植物转录组多样性和转录后修饰方面的独特优势。通过直接测序,研究人员能够更准确、更便捷地在一次实验中同时获取包含植物RNA序列与修饰位点在内的信息,为进一步探索不同条件下植物转录组的功能奠定基础。
此外,综述基于现有文献提出了一个系统的DRS数据分析流程,包括数据预处理、比对和生物学发现三个部分。数据预处理涉及对原始电信号的可视化与分析,以及从中提取碱基信息的base-calling。当前DRS reads的碱基错误率仍高于传统的RNA-seq,因此可以通过使用匹配组织的RNA-seq数据矫正DRS reads。校正后的DRS reads可与参考基因组或转录组比对,以获取全长转录本。最后通过定量分析、poly(A)长度估计和RNA碱基修饰检测等手段将DRS数据转换为有意义的生物学发现。此外,基于第三方性能评估、在植物研究中的使用频率以及实际应用的便利性,文章对一些核心分析步骤推荐了一系列工具,如Dorado用于Base-calling、FMLRC2用于reads校正、NanoCount用于转录本定量、TandemMod用于修饰碱基检测等(图2)。
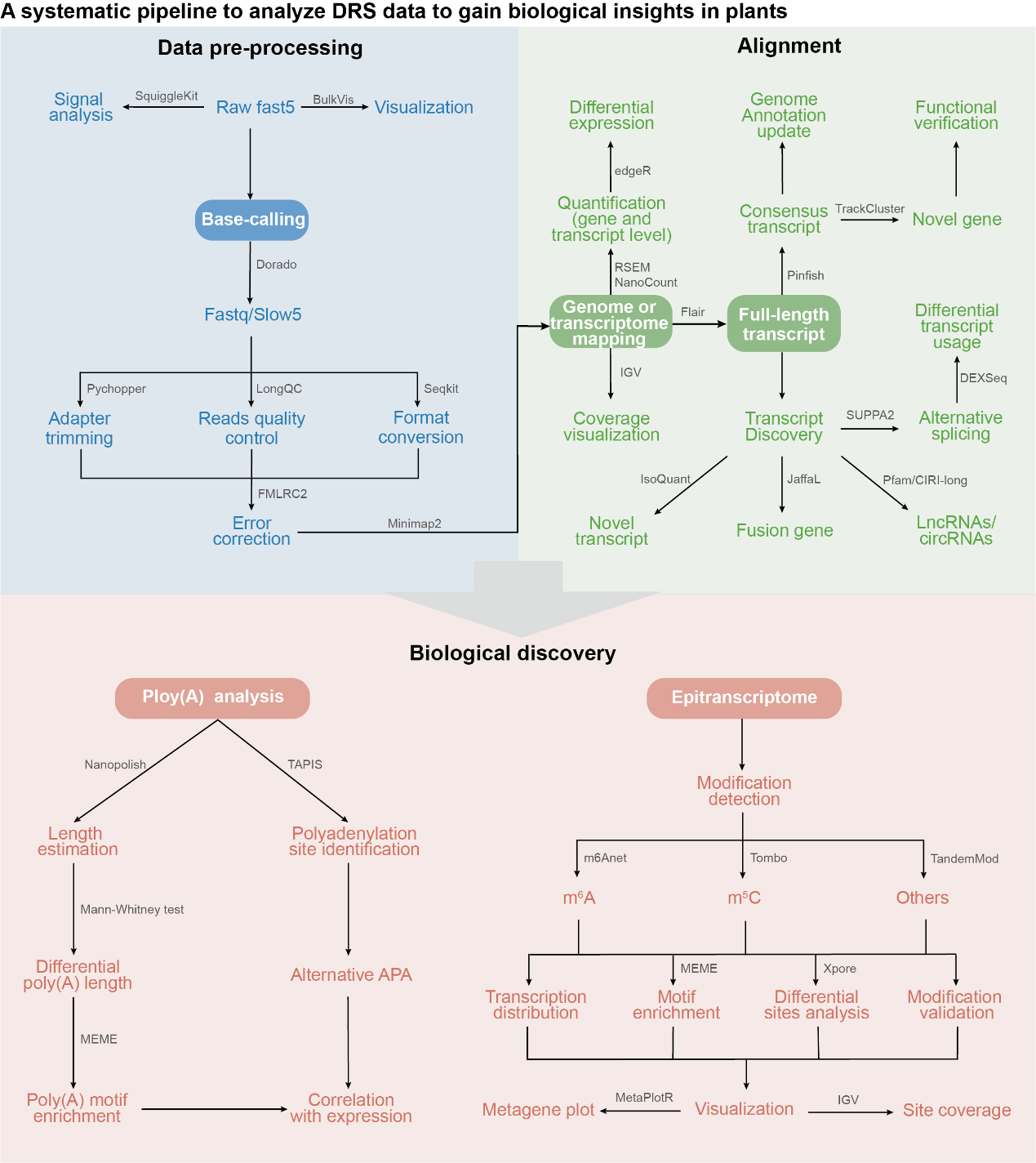
图2. DRS数据处理流程以及相应工具推荐.
尽管DRS技术带来了诸多机遇,但仍面临一系列挑战。DRS的碱基错误率和测序通量已经得到了大幅改善,但文库构建所需的RNA样本量仍远高于传统的RNA-seq以及direct cDNA sequencing,这限制了其在植物与微生物互作等领域中的应用。与此同时,尽管现有许多工具都能够用于处理DRS数据,但多数基于动物数据开发,专为植物设计的工具,尤其是深度学习方法,还相对匮乏。
随着测序成本的降低和通量的进一步提升,DRS 有望成为植物转录组学研究的主流工具,为揭示植物基因表达调控机制提供新的契机。其潜在的应用包括探索与转座元件有关转录本的功能、鉴定植物中真正的全长融合转录本、分析群体水平的RNA修饰及其与杂种优势的关系,以及揭示转录本异质性的单细胞RNA直接测序等。DRS技术不仅促进了植物生物学的进一步研究,还为作物改良和生物技术创新提供了新的可能性。
广西大学生命科学与技术学院博士后朱玺桐、硕士宁晓彤,华中农业大学信息学院Pablo为论文共同第一作者,广西大学陈玲玲教授和朱玺桐博士为论文的通讯作者。该项目得到了国家自然科学基金、广西科技重大专项等项目的支持。
文| 朱玺桐