甘蔗作为C4植物,在光合途径中具有高效的碳固定能力,其最高干物质产量可超过每公顷每年100干吨,全球80%的糖和40%的乙醇来自甘蔗。我国是全球第三大食糖生产国和第二大消费国,且90%以上的食糖来源于甘蔗。对甘蔗的研究不仅有助于深入理解C4植物的光合途径,还具有显著的农业应用潜力,能够提升作物产量。
尽管C4光合途径最早在甘蔗中被发现,但由于其复杂的遗传背景,甘蔗在分子水平上的C4光合途径研究进展相对缓慢。近年来,广西大学张积森教授团队对甘蔗C4光合代谢的分子机制进行了较系统的研究,取得了系列进展。团队揭示了甘蔗的C4光合特征,并解析了关键C4基因的调控机制(Zhang et al., 2018; Jiang et al., 2023; Hua et al., 2024)。此外,该团队还发现了一系列与光合作用相关的糖转运蛋白、镁离子转运蛋白和WRKY转录因子等(Hu et al., 2018; Li et al., 2019; Wang et al., 2019, 2022),为甘蔗的重要性状遗传改良提供分子依据。
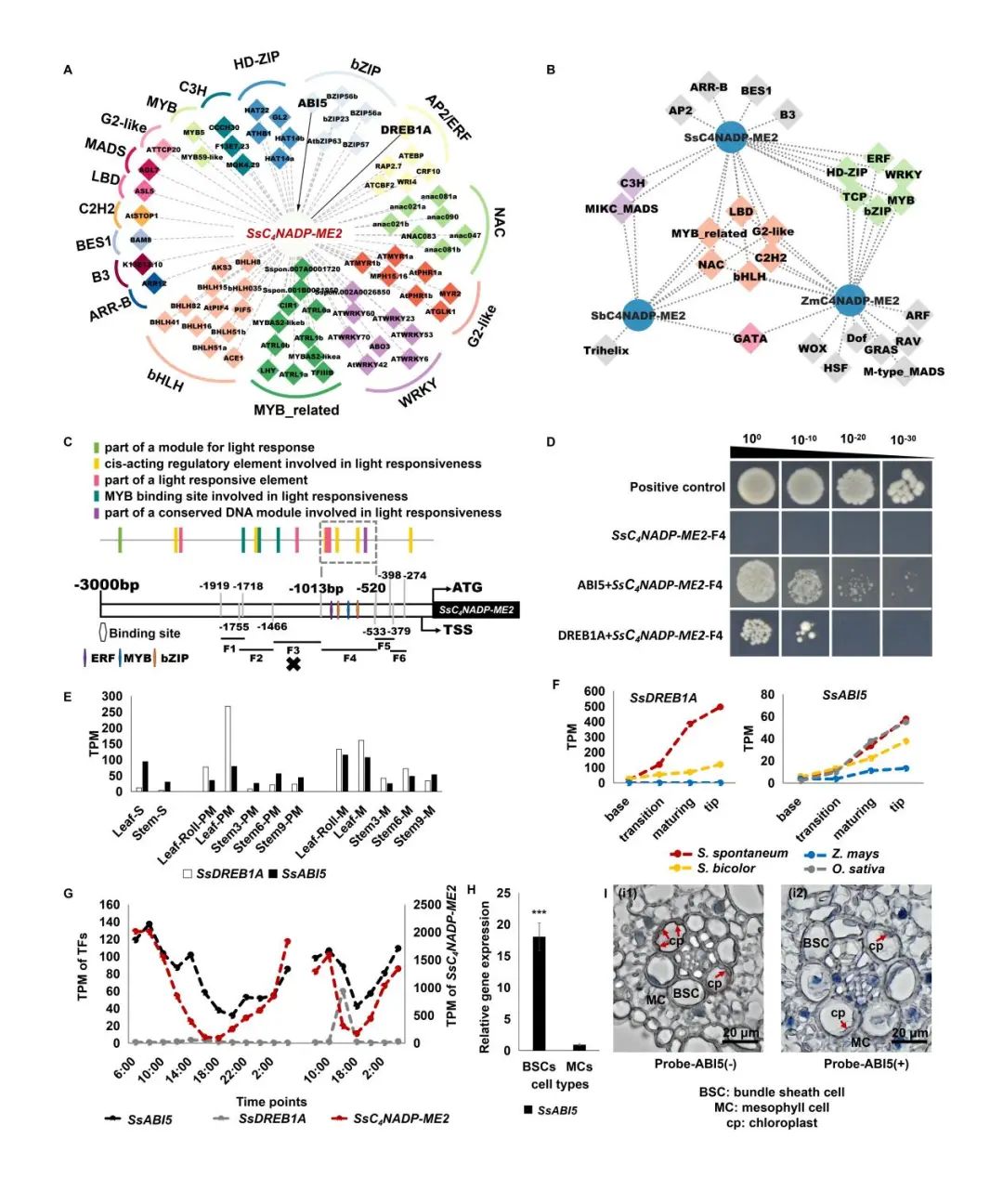
NADP苹果酸酶(C4NADP-ME)在叶片维管束鞘细胞中催化苹果酸的脱羧反应,释放CO2。在农业上重要的C4禾本科植物中,C4NADP-ME在C4光合途径中起着关键作用,促进了CO2的高效传输到Rubisco,从而影响了光合作用效率。由于甘蔗遗传背景复杂,研究其C4NADP-ME基因的分子调控面临挑战。
近日,张积森教授团队在Plant Physiology上发表了题为“Regulatory Network of the Late-Recruited Primary Decarboxylase C4NADP-ME in Sugarcane”的研究论文,揭示了甘蔗中后期招募的主要脱羧酶C4NADP-ME的调控网络。

研究显示,甘蔗割手密中的脱羧作用主要依赖于NADP-ME途径,类似于高粱和玉米。比较基因组学分析揭示了包括C4NADP-ME(SsC4NADP-ME2)在内的八个核心C4基因招募于甘蔗NADP-ME亚型C4途径中。与高粱和玉米不同,甘蔗中SsC4NADP-ME2的表达由不同的转录因子调控。研究构建了SsC4NADP-ME2的基因调控网络,并通过基因共表达分析和酵母单杂交实验等分子互作方法鉴定了候选转录因子。其中,ABI5被验证为调控SsC4NADP-ME2表达的主要候选转录因子,它结合在启动子区域的G-box内。值得注意的是,G-box中的核心元件ACGT在甘蔗、高粱、玉米和水稻中都高度保守,表明C4光合作用中利用了一种古老的调控机制。该研究为甘蔗作为生物能源作物的优化提供了宝贵的理论依据。
广西大学亚热带农业生物资源保护与利用国家重点实验室博士后华秀婷、福建农林大学海峡联合研究院基因组中心博士生石会红和广西大学亚热带农业生物资源保护与利用国家重点实验室博士生庄桂为论文共同第一作者。广西大学亚热带农业生物资源保护与利用国家重点实验室张积森教授、张木清教授、福建农林大学海峡联合研究院基因组中心唐海宝教授为论文的共同通讯作者。福建农林大学海峡联合研究院基因组中心明瑞光教授、广西大学亚热带农业生物资源保护与利用国家重点实验室姚伟教授、上海中科院植生所朱新广研究员、山东农业大学李平华教授等参与了本研究。同时,本研究还得到了广西大学金健教授、博士生余泽怀、鲍金林在实验上提供的的重要的指导和帮助。
主要参考文献:
1. Zhang J, Zhang X, Tang H, et al. Allele-defined genome of the autopolyploid sugarcane Saccharum spontaneum L. Nat Genet. 2018;50(11):1565-1573. doi:10.1038/s41588-018-0237-2
2. Hu W, Hua X, Zhang Q, et al. New insights into the evolution and functional divergence of the SWEET family in Saccharum based on comparative genomics. BMC Plant Biol. 2018;18(1):270. Published 2018 Nov 7. doi:10.1186/s12870-018-1495-y
3. Li Z, Hua X, Zhong W, et al. Genome-Wide Identification and Expression Profile Analysis of WRKY Family Genes in the Autopolyploid Saccharum spontaneum. Plant Cell Physiol. 2020;61(3):616-630. doi:10.1093/pcp/pcz227
4. Wang Y, Hua X, Xu J, et al. Comparative genomics revealed the gene evolution and functional divergence of magnesium transporter families in Saccharum. BMC Genomics. 2019;20(1):83. Published 2019 Jan 24. doi:10.1186/s12864-019-5437-3
5. Wang Y, Li Y, Hua X, et al. Transcriptome Dynamics Underlying Magnesium Deficiency Stress in Three Founding Saccharum Species. Int J Mol Sci. 2022;23(17):9681. Published 2022 Aug 26. doi:10.3390/ijms23179681
6. Jiang Q, Hua X, Shi H, et al. Transcriptome dynamics provides insights into divergences of the photosynthesis pathway between Saccharum officinarum and Saccharum spontaneum. Plant J. 2023;113(6):1278-1294. doi:10.1111/tpj.16110
7. Hua X, Li Z, Dou M, et al. Transcriptome and small RNA analysis unveils novel insights into the C4 gene regulation in sugarcane. Planta. 2024;259(5):120. Published 2024 Apr 12. doi:10.1007/s00425-024-04390-6