
小RNA广泛存在于植物中,对植物的生长发育和营养繁殖起至关重要的作用。目前已有多款植物小RNA分析工具,但大多数依赖于命令行环境,极大阻碍了湿实验室生物研究人员在植物小RNA生物功能挖掘上的深度探索。此外,当前的小RNA分析工具多集中在单一类型的小RNA分析上,且计算资源消耗较大,通常需要大量时间和精力来学习众多工具或脚本的使用并组成可用的流程以获得最终分析结果。
2023年12月30日,亚热带农业资源保护与利用国家重点实验室、华南农业大学夏瑞团队在Science Bulletin(IF:18.9)上在线发表了题为“sRNAminer: a Multifunctional Toolkit for Next-Generation Sequencing Small RNA Data Mining in Plants”的研究论文,该研究开发了一款多功能植物小RNA分析工具——sRNAminer。利用该工具,研究人员可进行一站式小RNA分析及可视化。
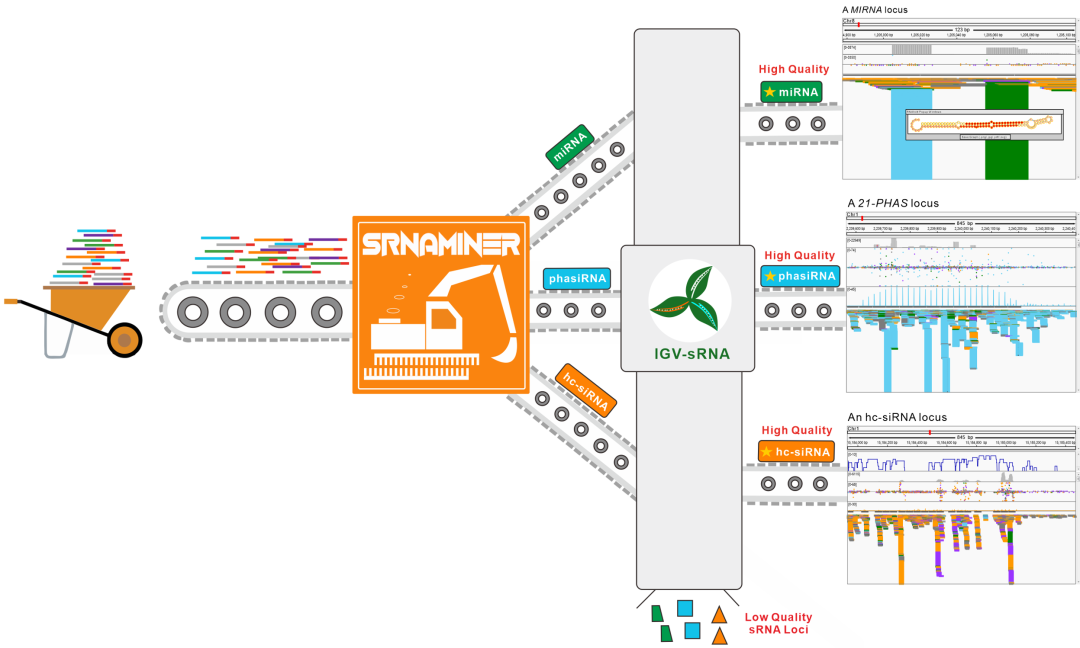
01 sRNAminer功能总览
sRNAminer包含了常用的植物小RNA数据分析功能。其功能主要分为四大部分:(1)数据预处理,包括接头去除,数据去冗余,去除目标外的非编码RNA(rRNA、tRNA、snoRNA、snRNA)和质体污染以及读段回帖。(2)植物三种主要类型小RNA【微RNA(microRNAs,miRNAs)、相位siRNAs(phased small interfering RNAs, phasiRNAs)和异染色质siRNAs(heterochromatic siRNAs,hc-siRNAs)】的鉴定与表达量计算。(3)其他常用的功能包括:小RNA靶位点预测,介导PHAS位点产生的小RNA trigger预测,降解组测序数据分析等等(图1a)。(4)小RNA位点可视化。sRNAminer的IGV-sRNA模块让用户能够针对相应类型的小RNA的特征对小RNA位点进行快速人工检查。
为保证用户能够以最快最简单的方式进行小RNA分析,sRNAminer提供了一键化分析功能。用户只需要输入小RNA测序文件、基因组文件以及软件配备的公共非编码RNA(rRNA、tRNA、snoRNA、snRNA)库和质体基因组数据库,点击“Start”按钮,软件便会自动解析数据,对数据进行预处理,将读段回贴到基因组,然后进行小RNA鉴定,最终在较短时间内即可得到miRNA、phasiRNA和hc-siRNA的鉴定结果及其表达量(图1b和2)。
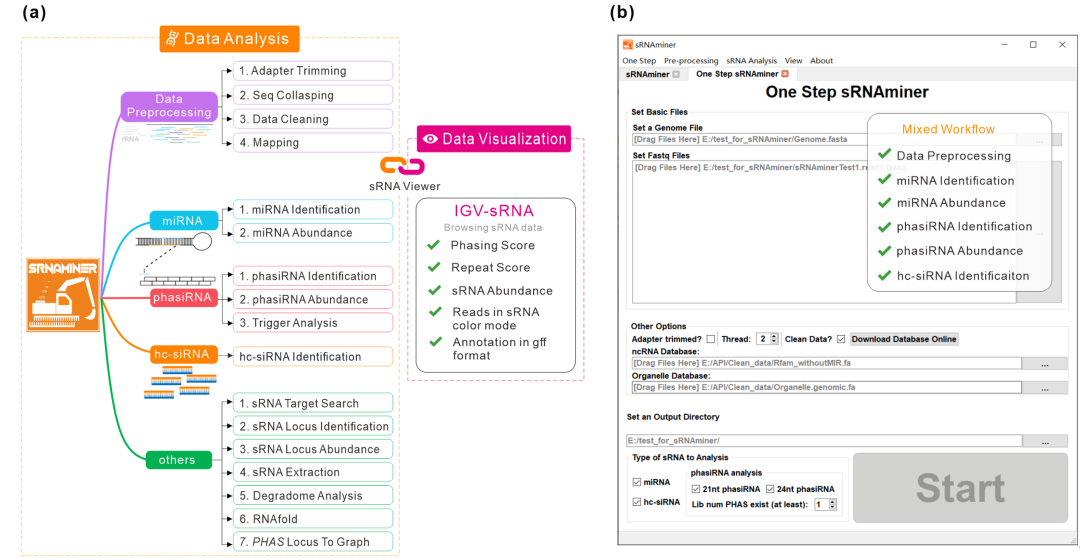
图1 sRNAminer功能概览
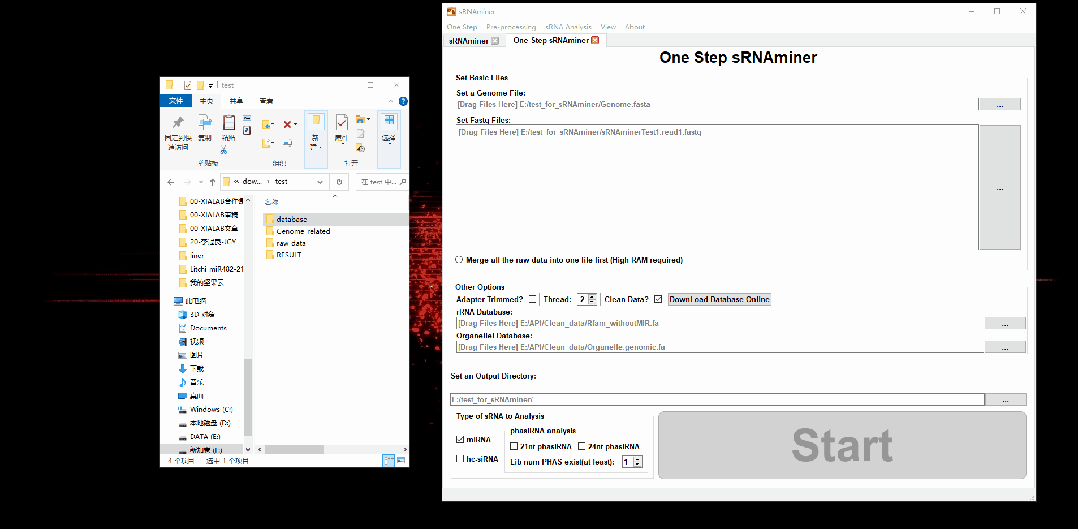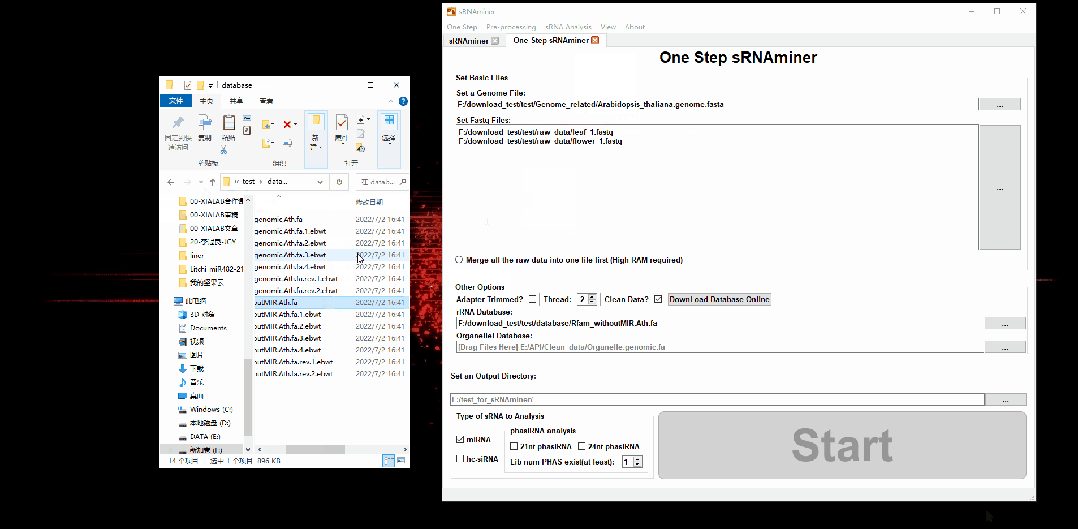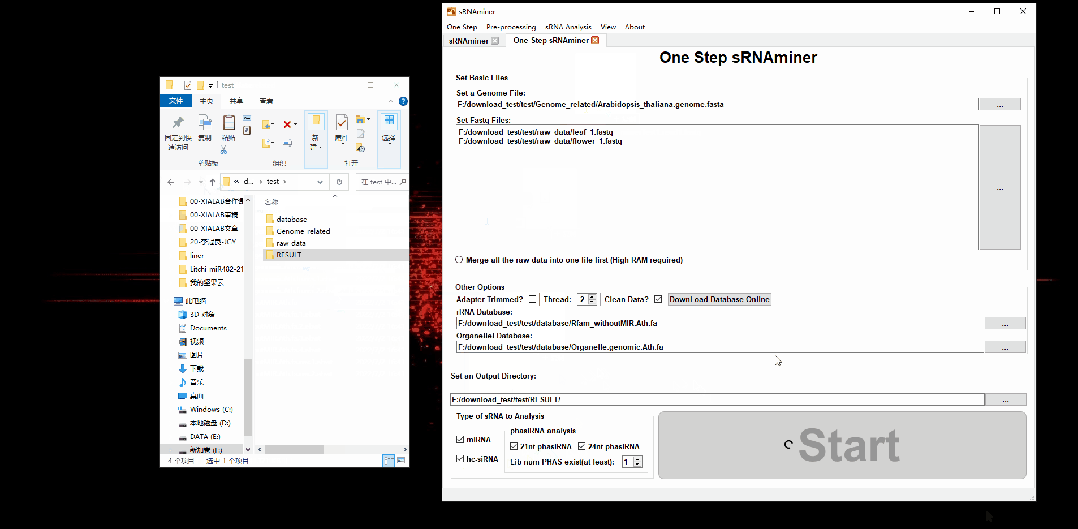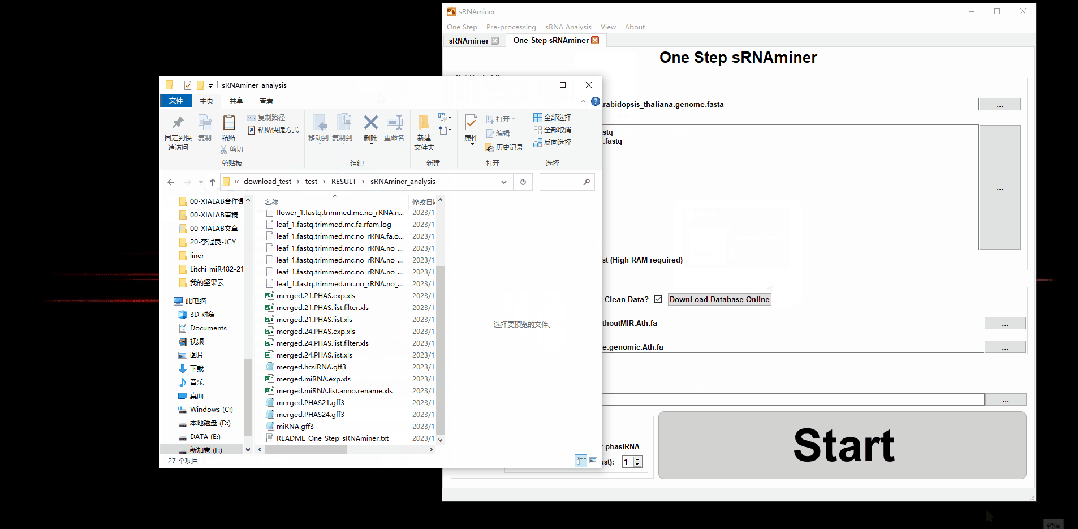
图2 “One Step sRNAminer”功能演示
sRNAminer兼具界面化和命令行版本。在界面化版本下,用户只需要“拖拖拖,点点点“即可完成多种类型的小RNA分析(图2),大大降低了小RNA分析的门槛。针对拥有大基因组(>3Gb)的物种及大型数据集,本研究为用户提供sRNAminer的命令行版本(图3),使其能够在高性能的计算服务器上使用sRNAminer进行小RNA分析。

图3 sRNAminer命令行版本操作示例
02 sRNAminer提供快速且准确的小RNA注释
为了更客观地评价不同软件的表现,本研究经过比较分析和人工检查构建了一个高标准的“黄金”MIRNA和PHAS位点集合(图4),用于评估sRNAminer与其他现有工具的性能。在miRNA鉴定方面,本研究比较了不同软件在拟南芥和水稻上的表现。结果显示sRNAminer拥有优于/不亚于其他软件的分析结果,并且在分析时间上比排名第二的miRDeep-P2快了一倍(图5)。在PHAS位点鉴定方面,本研究将sRNAminer与PhaseTank进行比较。结果显示,sRNAminer能够比PhaseTank鉴定到更多的高可信PHAS位点(图6)。
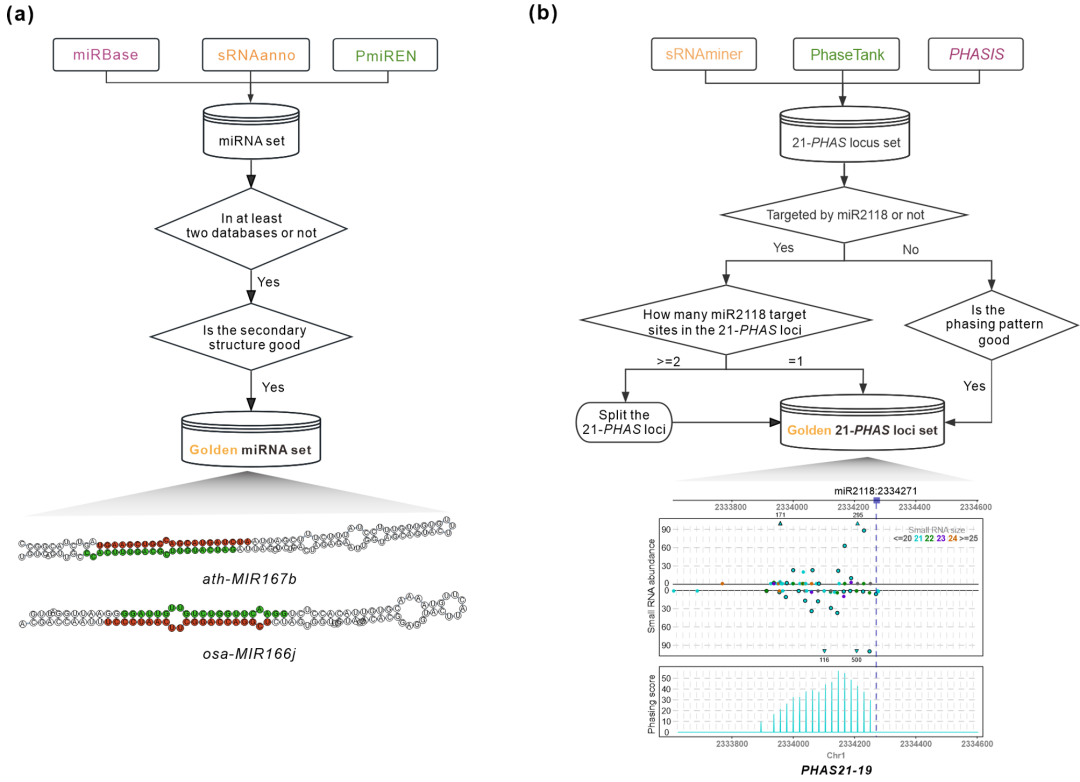
图4 “黄金”MIRNA和PHAS位点集合的构建
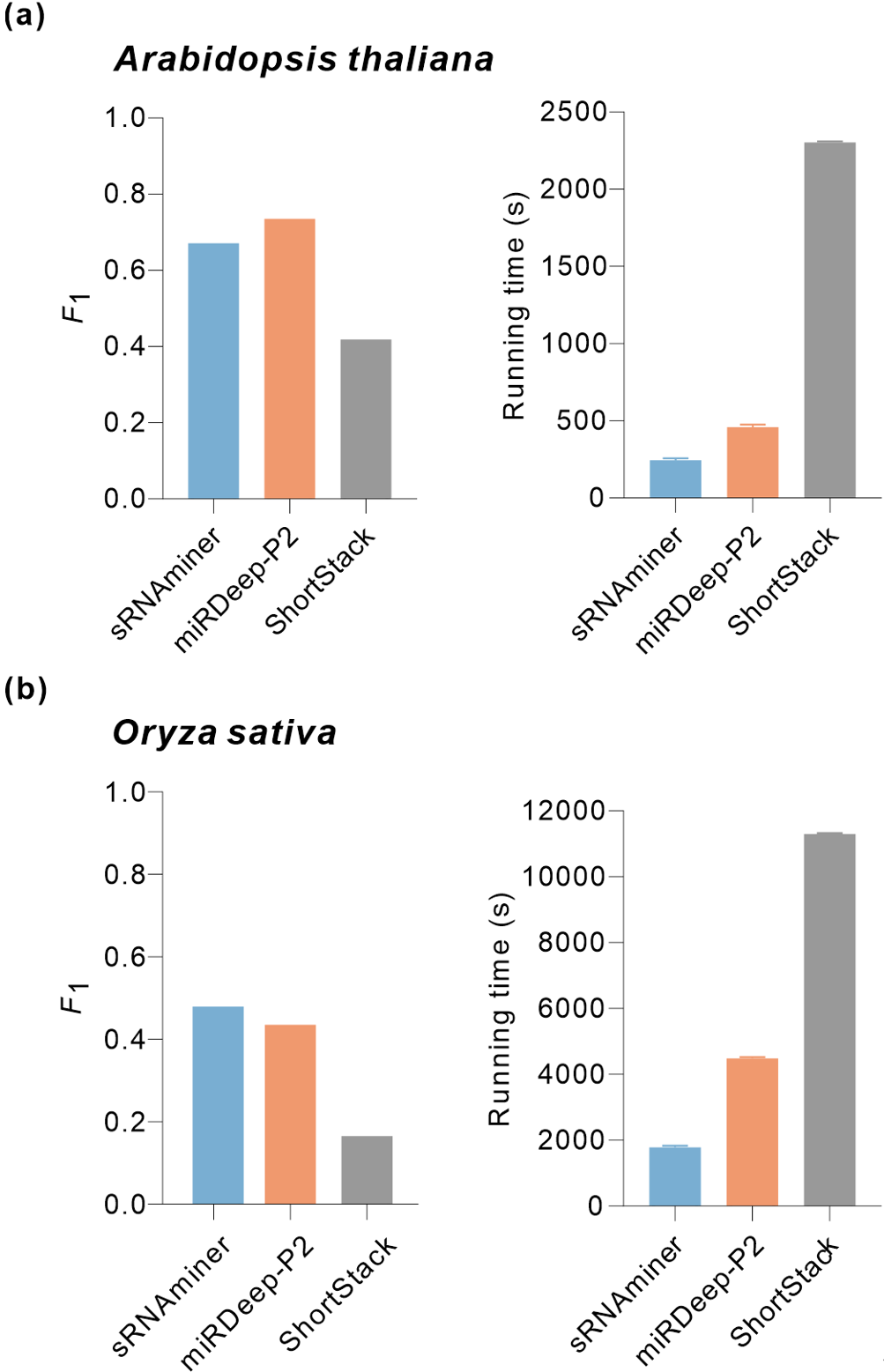
图5 sRNAminer与其他软件在miRNA鉴定上的表现比较
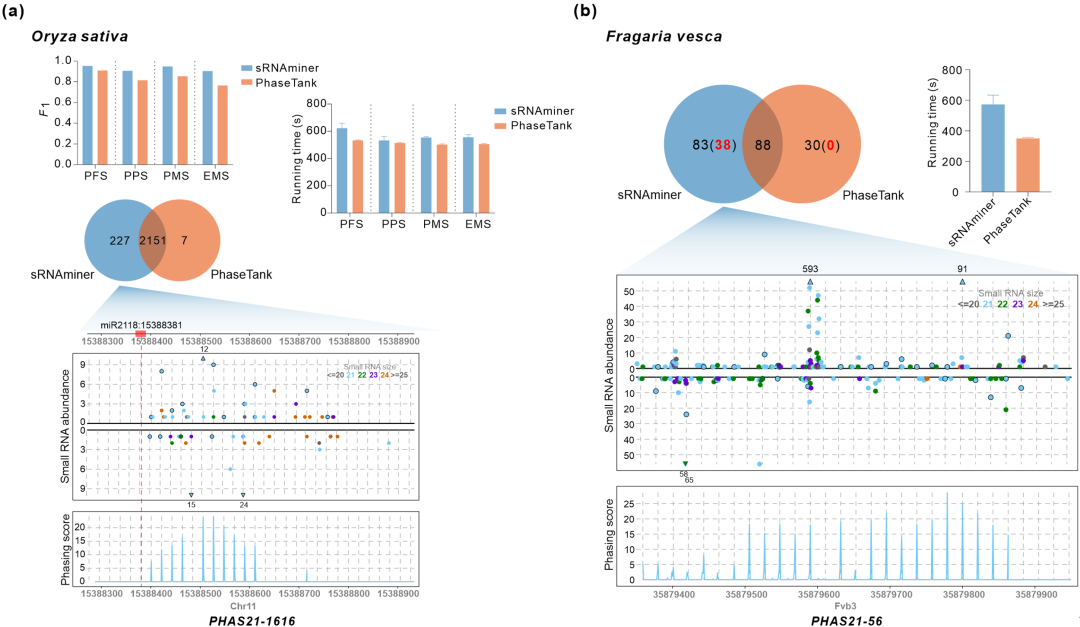
图6 sRNAminer与PhaseTank在PHAS位点鉴定上的表现比较
03 sRNAminer可视化板块
为了实现对小RNA注释结果的高效人工检查,本研究基于基因组浏览器(IGV)开发了IGV-sRNA,并将其作为一个功能模块整合在sRNAminer里。IGV-sRNA可以呈现各种特异的小RNA特征,使研究人员能够更全面地理解小RNA数据(图7)。另外,用户配合sRNAminer的“sRNA Viewer”功能,即可快速跳转到对应的小RNA位点上,从而高效地查看小RNA位点(图8)。此外,sRNAminer支持高质量PHAS位点图批量自动绘制及RNA二级结构可视化(图9)。sRNAminer可以真正实现从分析数据到人工检查可视化结果再到高质量图表绘制的小RNA分析流程闭环。
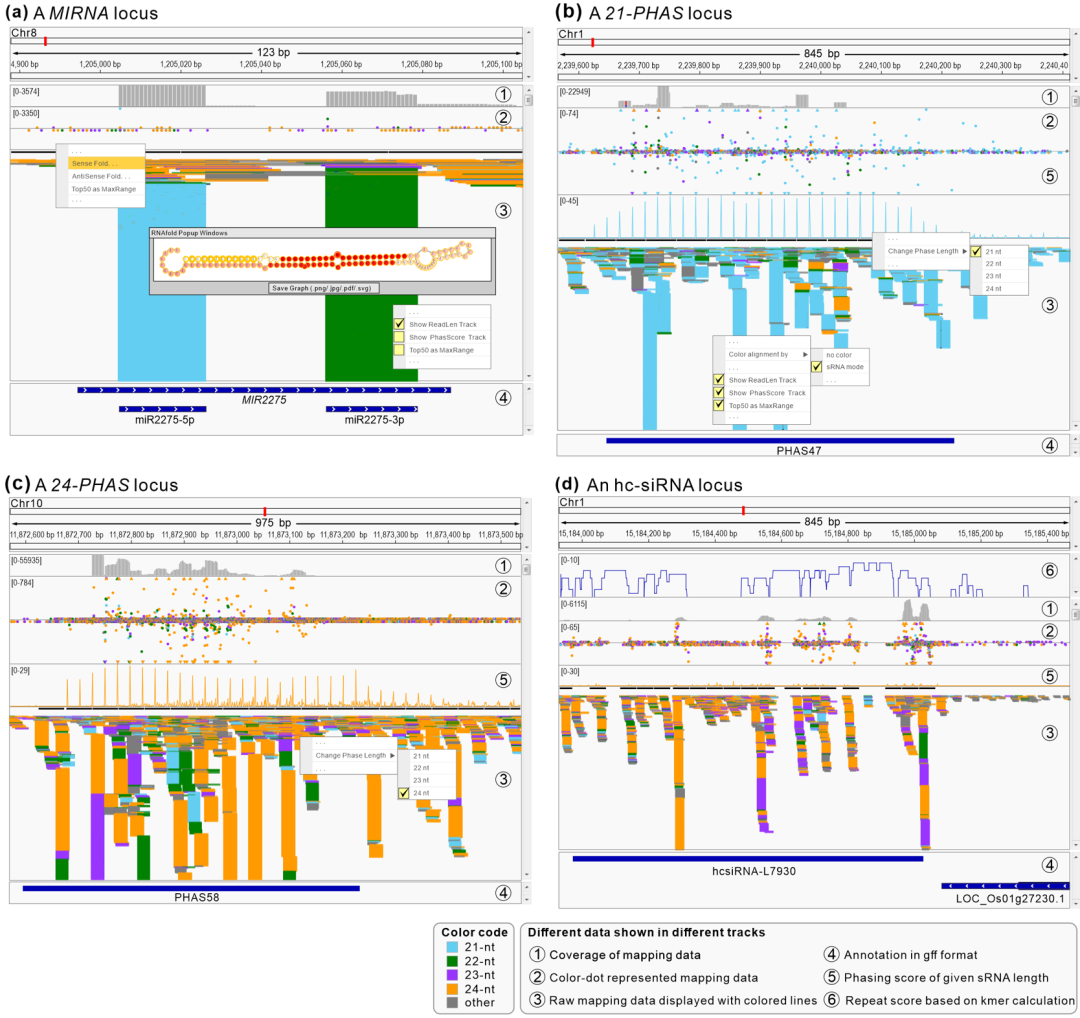
图7 IGV-sRNA功能概览
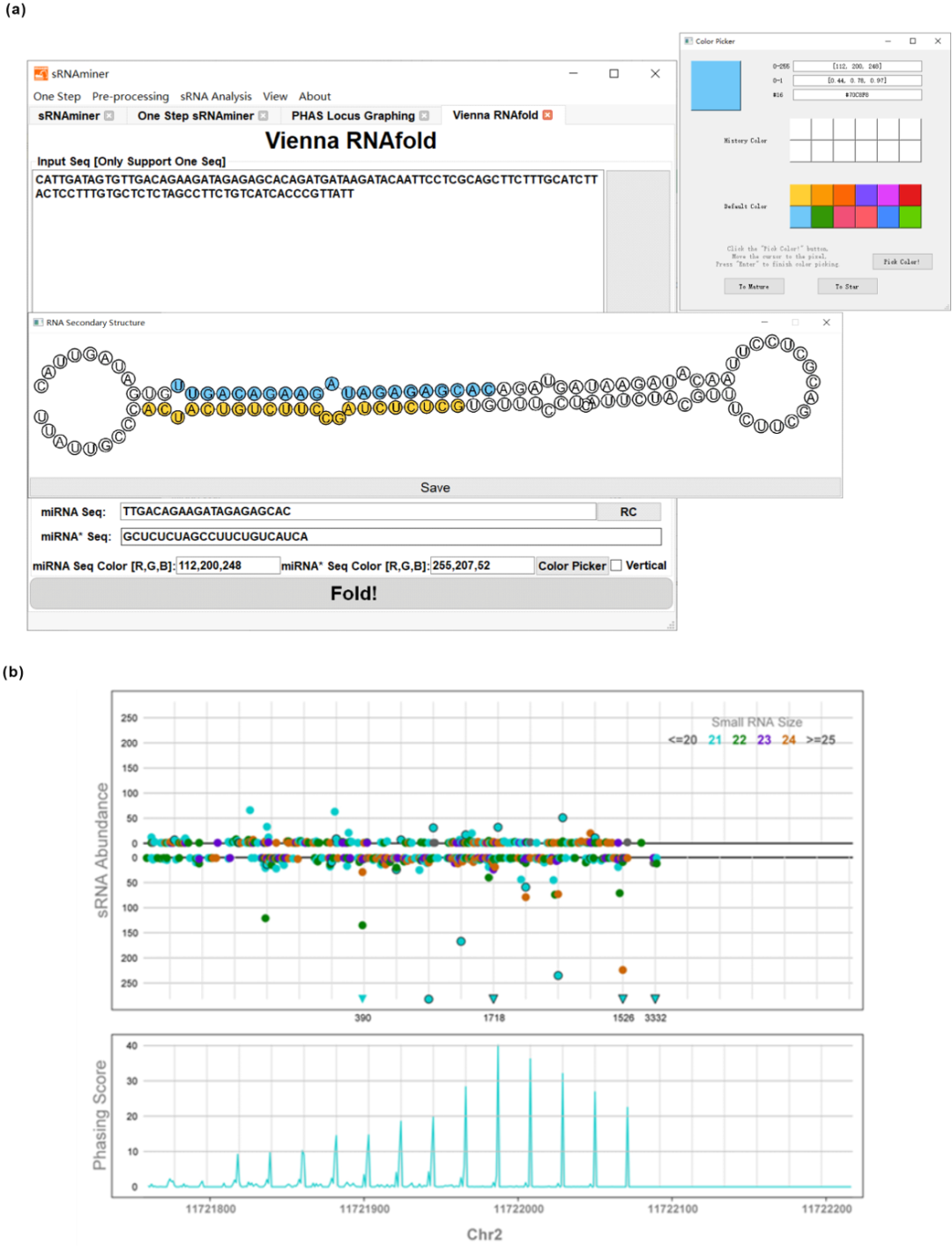
图8 “sRNA Viewer”功能展示

图9 sRNAminer的RNA二级结构可视化和高质量PHAS位点图绘制结果
sRNAminer和IGV-sRNA均为跨平台软件,可以在多个操作系统下运行。安装包可在https://github.com/kli28/sRNAminer和https://gitee.com/CJchen/IGV-sRNA免费下载。使用手册在https://www.yuque.com/u758713/at2327。
本论文以华南农业大学为第一完成单位,华南农业大学园艺学院夏瑞教授、陈程杰博士为共同通讯作者。已毕业硕士研究生李冠良和陈程杰博士为该论文共同第一作者。硕士研究生陈佩珂参与了该论文的数据分析。美国唐纳德丹佛斯植物科学中心Blake C. Meyers教授对该研究做出了贡献。该研究得到国家自然科学基金、广东省重点研发项目,海南崖州湾种子实验室等资助。